【生物信息学】使用HSIC LASSO方法进行特征选择
目录
一、实验介绍
二、实验环境
1. 配置虚拟环境
2. 库版本介绍
3. IDE
三、实验内容
0. 导入必要的工具
1. 读取数据
2. 划分训练集和测试集
3. 进行HSIC LASSO特征选择
4. 特征提取
5. 使用随机森林进行分类(使用所有特征)
6. 使用随机森林进行分类(使用HSIC选择的特征):
7. 代码整合
一、实验介绍
本实验实现了HSIC LASSO(Hilbert-Schmidt independence criterion LASSO)方法进行特征选择,并使用随机森林分类器对选择的特征子集进行分类。
特征选择是机器学习中的重要任务之一,它可以提高模型的效果、减少计算开销,并帮助我们理解数据的关键特征。
HSIC LASSO是一种基于核的独立性度量方法,用于寻找对输出值具有强统计依赖性的非冗余特征。
二、实验环境
本系列实验使用了PyTorch深度学习框架,相关操作如下(基于深度学习系列文章的环境):
1. 配置虚拟环境
深度学习系列文章的环境
conda create -n DL python=3.7 conda activate DLpip install torch==1.8.1+cu102 torchvision==0.9.1+cu102 torchaudio==0.8.1 -f https://download.pytorch.org/whl/torch_stable.html
conda install matplotlibconda install scikit-learn新增加
conda install pandasconda install seabornconda install networkxconda install statsmodelspip install pyHSICLasso注:本人的实验环境按照上述顺序安装各种库,若想尝试一起安装(天知道会不会出问题)
2. 库版本介绍
| 软件包 | 本实验版本 | 目前最新版 |
| matplotlib | 3.5.3 | 3.8.0 |
| numpy | 1.21.6 | 1.26.0 |
| python | 3.7.16 | |
| scikit-learn | 0.22.1 | 1.3.0 |
| torch | 1.8.1+cu102 | 2.0.1 |
| torchaudio | 0.8.1 | 2.0.2 |
| torchvision | 0.9.1+cu102 | 0.15.2 |
新增
| networkx | 2.6.3 | 3.1 |
| pandas | 1.2.3 | 2.1.1 |
| pyHSICLasso | 1.4.2 | 1.4.2 |
| seaborn | 0.12.2 | 0.13.0 |
| statsmodels | 0.13.5 | 0.14.0 |
3. IDE
建议使用Pycharm(其中,pyHSICLasso库在VScode出错,尚未找到解决办法……)
win11 安装 Anaconda(2022.10)+pycharm(2022.3/2023.1.4)+配置虚拟环境_QomolangmaH的博客-CSDN博客https://blog.csdn.net/m0_63834988/article/details/128693741![]() https://blog.csdn.net/m0_63834988/article/details/128693741
https://blog.csdn.net/m0_63834988/article/details/128693741
三、实验内容
0. 导入必要的工具
import random
import pandas as pd
from pyHSICLasso import HSICLasso
from sklearn.preprocessing import LabelEncoder
from sklearn.model_selection import train_test_split
from sklearn.metrics import confusion_matrix, classification_report
from sklearn.ensemble import RandomForestClassifier1. 读取数据
data = pd.read_csv("cancer_subtype.csv")
x = data.iloc[:, :-1]
y = data.iloc[:, -1]2. 划分训练集和测试集
X_train, X_test, y_train, y_test = train_test_split(x, y, test_size=0.3, random_state=10) 将数据集划分为训练集(X_train和y_train)和测试集(X_test和y_test)。其中,测试集占总数据的30%。
3. 进行HSIC LASSO特征选择
random.seed(1)
le = LabelEncoder()
y_hsic = le.fit_transform(y_train)
x_hsic, fea_n = X_train.to_numpy(), X_train.columns.tolist()
hsic.input(x_hsic, y_hsic, featname=fea_n)
hsic.classification(200)
genes = hsic.get_features()
score = hsic.get_index_score()
res = pd.DataFrame([genes, score]).T- 设置随机种子,确保随机过程的可重复性
- 使用
LabelEncoder将目标变量进行标签编码,将其转换为数值形式。 - 通过将训练集数据
X_train和标签y_hsic输入HSIC LASSO模型进行特征选择。hsic.input用于设置输入数据和特征名称hsic.classification用于运行HSIC LASSO算法进行特征选择- 选择的特征保存在
genes中; - 对应的特征得分保存在
score中;
- 选择的特征保存在
- 将
genes、score存储在DataFrameres中。
4. 特征提取
hsic_x_train = X_train[res[0]]
hsic_x_test = X_test[res[0]] 根据HSIC LASSO选择的特征索引,从原始的训练集X_train和测试集X_test中提取相应的特征子集,分别保存在hsic_x_train和hsic_x_test中。
5. 使用随机森林进行分类(使用所有特征)
rf_model = RandomForestClassifier(20)
rf_model.fit(X_train, y_train)
rf_pred = rf_model.predict(X_test)
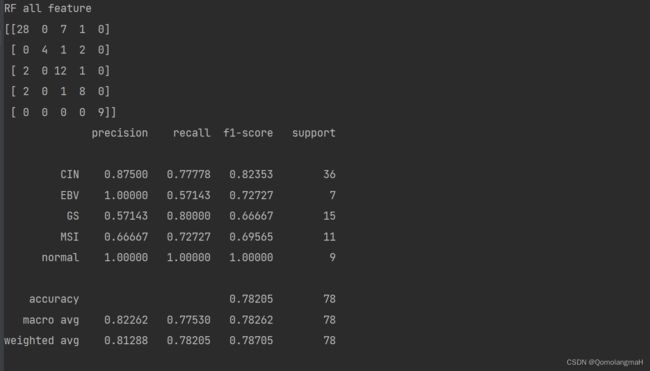
print("RF all feature")
print(confusion_matrix(y_test, rf_pred))
print(classification_report(y_test, rf_pred, digits=5)) 使用随机森林分类器(RandomForestClassifier)对具有所有特征的训练集进行训练,并在测试集上进行预测。预测结果存储在rf_pred中,并输出混淆矩阵(confusion matrix)和分类报告(classification report)。
6. 使用随机森林进行分类(使用HSIC选择的特征):
rf_hsic_model = RandomForestClassifier(20)
rf_hsic_model.fit(hsic_x_train, y_train)
rf_hsic_pred = rf_hsic_model.predict(hsic_x_test)
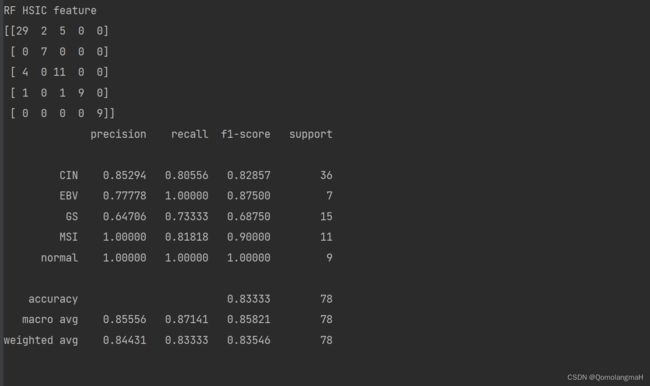
print("RF HSIC feature")
print(confusion_matrix(y_test, rf_hsic_pred))
print(classification_report(y_test, rf_hsic_pred, digits=5)) 使用随机森林分类器对使用HSIC LASSO选择的特征子集hsic_x_train进行训练,并在测试集的相应特征子集hsic_x_test上进行预测。预测结果存储在rf_hsic_pred中,并输出混淆矩阵和分类报告。
7. 代码整合
# HSIC LASSO
# HSIC全称“Hilbert-Schmidt independence criterion”,“希尔伯特-施密特独立性指标”,跟互信息一样,它也可以用来衡量两个变量之间的独立性
# 核函数的特定选择,可以在基于核的独立性度量(如Hilbert-Schmidt独立性准则(HSIC))中找到对输出值具有很强统计依赖性的非冗余特征
# CIN 107 EBV 23 GS 50 MSI 47 normal 33
import random
import pandas as pd
from pyHSICLasso import HSICLasso
from sklearn.preprocessing import LabelEncoder
from sklearn.model_selection import train_test_split
from sklearn.metrics import confusion_matrix, classification_report
from sklearn.ensemble import RandomForestClassifier
data = pd.read_csv("cancer_subtype.csv")
x = data.iloc[:, :-1]
y = data.iloc[:, -1]
X_train, X_test, y_train, y_test = train_test_split(x, y, test_size=0.3, random_state=10)
random.seed(1)
le = LabelEncoder()
hsic = HSICLasso()
y_hsic = le.fit_transform(y_train)
x_hsic, fea_n = X_train.to_numpy(), X_train.columns.tolist()
hsic.input(x_hsic, y_hsic, featname=fea_n)
hsic.classification(200)
genes = hsic.get_features()
score = hsic.get_index_score()
res = pd.DataFrame([genes, score]).T
hsic_x_train = X_train[res[0]]
hsic_x_test = X_test[res[0]]
rf_model = RandomForestClassifier(20)
rf_model.fit(X_train, y_train)
rf_pred = rf_model.predict(X_test)
print("RF all feature")
print(confusion_matrix(y_test, rf_pred))
print(classification_report(y_test, rf_pred, digits=5))
rf_hsic_model = RandomForestClassifier(20)
rf_hsic_model.fit(hsic_x_train, y_train)
rf_hsic_pred = rf_hsic_model.predict(hsic_x_test)
print("RF HSIC feature")
print(confusion_matrix(y_test, rf_hsic_pred))
print(classification_report(y_test, rf_hsic_pred, digits=5))