- 900 万人次都在用!打印机驱动大师:兄弟 驱动安装一步到位
文哥工具箱2
软件工程电脑开源软件
各位打印界的老铁们,你们知道吗?我就是那个传说中服务PT-18R标签打印机的“最佳损友”小助手!当你想把电脑里那些花里胡哨的标签设计变成能摸得着的实物时,嘿嘿,软件下载地址本助手就闪亮登场啦!插上USB线的瞬间,我立马在你电脑里“安营扎寨”,悄悄给你和打印机搭起一座“鹊桥”,让你们无障碍沟通,那叫一个丝滑!你在编辑软件里鼓捣的文字、条形码,甚至那些可可爱爱的小图标,全靠我这个“翻译官”精准转换成打
- Anaconda 和 Miniconda:功能详解与选择建议
古月฿
python入门pythonconda
Anaconda和Miniconda详细介绍一、Anaconda的详细介绍1.什么是Anaconda?Anaconda是一个开源的包管理和环境管理工具,在数据科学、机器学习以及科学计算领域发挥着关键作用。它以Python和R语言为基础,为用户精心准备了大量预装库和工具,极大地缩短了搭建数据科学环境的时间。对于那些想要快速开展数据分析、模型训练等工作的人员来说,Anaconda就像是一个一站式的“数
- 2/7-1组-泡菜
上海健康产业蔡文俊
片段主题:分享、分享、再分享片段来源:认知突围R:很多人并不清楚,其实分享是最好的学习方法。第一,分享是一件利己利人的事。我把好的思想或方法论分享给你,你多少有所得。反过来,也许你会提出一些不同的东西,触发我更多的思考。哪怕你并没有提出什么新见解,单单是我自己在分享的时候,也是一个对知识的自我记忆和强化的过程,依然是非常有好处的。第二,分享是一件能倒逼你完善知识体系的事。很多人都有过这样的经历,当
- 宝妈兼职的10个项目这些较适合,这10个适合长期发展的正规兼职请收藏
氧惠全网优惠
大家好!我是氧惠平台最大团队&联合创始人破局导师。相较于其它返利app,氧惠佣金更高,模式更好,终端用户不流失!氧惠目前属于邀请制社交电商,需要凭邀请码才能体验,请输入我的邀请码:SJ8M8R,进行体验吧!宝妈兼职是一种越来越受欢迎的工作方式,适用于压力山大的家庭主妇、退休妇女以及需要额外收入的人们。这种工作方式可以实现财务自由和工作生活平衡。下面将为大家介绍10种适合宝妈兼职的正规长期项目。第一
- R语言笔记Day1(排序、筛选以及分类汇总))
养猪场小老板
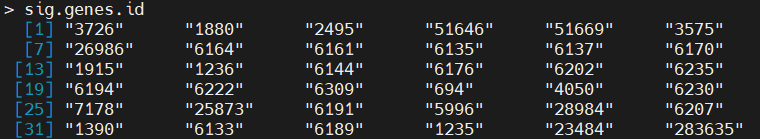
一、排序1、单变量序列排序2、数据表(矩阵)排序二、筛选三、分类汇总一、排序1、单变量序列排序rank、sort和order函数>aa[1]315#rank用来计算序列中每个元素的秩#这里的“秩”可以理解为该元素在序列中由小到大排列的次序#上面例子给出的序列[3,1,5]中,1最小,5最大,3居中#于是1的秩为1,3的秩为2,5的秩为3,(3,1,5)对应的秩的结果就是(2,1,3)>rank(a
- 2022年拼多多618活动什么时候开始和结束
日常购物技巧呀
马上就是618咯,小伙们准备好购物资金了吗?可不能将鸡蛋都放在一个篮子里,多去各个电商平台逛逛,享受多重618优惠服务。那么2022拼多多618活动什么时候开始呢?快和小编一起来了解一下吧。时间:5.29日20点—6.20日每天抽三次红包,最高可领28888r‼️参与方式很简单,直接去桃宝搜官方密令即可领取,现在分享给大家!密令:【天降红包61666】这是官方密令,中大包的概率更高,现在就可以去搜
- Linux文件权限与进程管理解析
雨季西柚
linux
控制对文件的访问1。什么是文件系统权限?它是如何工作的?如何查看文件的权限?答:文件系统权限就是规定谁能对文件/文件夹做什么(比如看、改、删)的规则。简单说,就是分"所有者、所属组、其他人"三类,给每类分配"读、写、执行"三种权限。操作时系统先看你属于哪类,再查有没有对应权限,有就允许,没有就拦着。查看方式:Linux/mac:终端输ls-l文件名,看开头的rwxr--r--这类字符(3个一组,对
- 智慧施工:AI技术赋能建筑安全监测新纪元
开发AI智能应用,就下载InsCodeAIIDE,一键接入DeepSeek-R1满血版大模型!智慧施工:AI技术赋能建筑安全监测新纪元在现代建筑行业中,施工安全始终是核心关注点之一。随着科技的飞速发展,人工智能(AI)和大数据分析逐渐成为提升施工安全的重要工具。本文将探讨如何利用智能化软件和大模型API来构建高效的施工安全监测系统,并介绍一款强大的开发工具——InsCodeAIIDE的应用场景及其
- 控制障碍函数
控制障碍函数(ControlBarrierFunction,CBF)是控制理论中的一个重要工具,尤其是在确保系统安全和稳定性方面。CBF使得控制系统能够在运行的同时遵循特定的约束条件,从而避免进入不安全的状态。接下来,我们将详细介绍控制障碍函数的定义、性质及其应用。1.控制障碍函数的定义控制障碍函数是用于描述系统状态在某些约束条件下的行为的函数。通常,一个控制障碍函数b:Rn→R通过以下形式定义约
- MySQL笔记上
程程呀是小白
毕业季mysql数据库
本文是本人以前笔记,如果说是在掘金上看到的话没错,还是本人程程呀的个人主页-动态-掘金只有这一个。如果有哪里不对的话欢迎各位大佬指出问题,本人是一个小白。特殊情况(倘若在终端win+r输入mysql-uroot-p进入SQL)MySQL的下载与安装:按照安装流程步骤安装即可配置环境变量,方便命令行操作:步骤:右键“我的电脑”,选择“属性”,选择“高级”窗口中的“环境变量”,设置“系统变量”中的pa
- 阿里云企业新用户与个人新用户首次购买云服务器配置推荐
阿里云最新优惠和活动汇总
2023年阿里云活动中的云服务器实例规格以通用算力型u1、计算型c7、通用型g7、内存型r7等为主,但是阿里云将各活动中的云服务器可选带宽统一调整成为了1-5M可选,也就是说在活动中购买云服务器最大只能选5M带宽了,活动时长也由以往的1-3年改为现在的1年为主,根据目前的活动信息,小编决定为大家推荐两款值得企业新用户与个人新用户首次购买时选择的阿里云服务器。阿里云个人新用户首次购买云服务器推荐推荐
- Error: C++14 standard requested but CXX14 is not defined
闹钟又响了
在R中安装包如果遇到上述问题,基本上去修改一下Makevars文件即可。可以用vim编辑,也可以在R中编辑,如下dotR<-file.path(Sys.getenv("HOME"),".R")if(!file.exists(dotR))dir.create(dotR)M<-file.path(dotR,"Makevars")if(!file.exists(M))file.create(M)cat(
- 一对多/多对一RIA
李健Brian
【概念/关键词名称】一对多、多对一【分类】[x]wow突破点[x]知识连接点[x]探索点【R:原文】【多对一:多模型思维】*所谓科学的方法主要体现为“可复现”。也就是说,只要定理假设的条件满足了,多模型思维就一定会比单模型思维好,不以任何外在因素而变化。这也是模型的威力所在。*【孔多赛陪审团定理】*陪审团是一种通过多数投票进行决策的方式,一般是二元决策——有罪或无罪,陪审团成员之间彼此不受影响地独
- 沿瑾+教练春哥+第三拆作业
sunny0902
拆书帮长沙橘洲分舵第10期训练营第三拆作业~2020.03.18选自《亲密关系》P163-164【R•阅读原文片段】在沟通中当我们接收到他人的信息时,有两个重要的任务要完成。第一是要准确的理解对方话语所表达的意思,第二是要向对方传达关注和理解,让他知道我们对他的话是在意的。这两个任务都可以通过复述(paraphrasing)接受到的信息而完成。即用自己的话重复对方的意思,让信息传递着有机会肯定那就
- 远程服务器windows对外开放端口,Windows Server 2008 R2开启新端口远程桌面
【要求】1、修改默认端口。2、不用关闭防火墙。【步骤】1、用PowerShell查看:(Get-ItemProperty-Path"hklm:SYSTEM\CurrentControlSet\Control\TerminalServer\Wds\rdpwd\Tds\tcp").PortNumber(Get-ItemProperty-Path"hklm:SYSTEM\CurrentControlSe
- 10月26日分享
移动小翁
A:计划赶不上变化,上午8点半接到分管领导电话,9点宁德地区三级经理9人,临时要加入队伍开展行销。M:紧张,忐忑。B:调整心态,马上协商本次罗源支援团体的市场部经理,分配人员行销扫街路线图及分组。通过罗源及宁德公司人员混搭,带领他们学习福州扫街的模式。R:宁德公司对我们的工作认可,同时学习到了福州公司行销的模式。
- 从0开始学习R语言--Day58--竞争风险模型
在用传统生存分析方法的场景中(如Kaplan-Meier和Cox回归),假设所有事件都是独立且互斥的,但在现实中,研究对象可能面临多种互斥的终点事件(如癌症患者可能死于癌症本身,也可能死于其他原因),如果直接去分析,模型会把这种结局时间错误地纳入评估,从而提高了病症的分析。而竞争风险模型可以在考虑其他竞争风险存在的情况下,排除干扰求得某特定事件发生的概率。以下是一个例子:library(cmprs
- day18-作业
oct___d38e9
server.pyfromsocketimport*importosserver=socket()server.bind(('10.7.181.100',8888))server.listen(512)withopen('start.txt','r',encoding='utf-8')asf:start=f.read()whileTrue:connet,adress=server.accept()
- MGRE综合实验报告
MGRE综合实验实验步骤:1.先写DHCP服务(动态主机配置协议)私有网段AR1-4对应内网的不冲突设计R1-R4的DHCP和网关配置,写完代码,打开PC基础配置将静态改为DHCP[r1]dhcpenable[r1]ippool1[r1-ip-pool-1]network192.168.1.0mask24[r1-ip-pool-1]gateway-list192.168.1.254[r1-ip-p
- 强化学习入门三(SARSA)
第六五签
算法模型算法人工智能
SARSA算法详解SARSA是强化学习中另一种经典的时序差分(TD)学习算法,与Q-Learning同属无模型(model-free)算法,但在更新策略上有显著差异。SARSA的名称来源于其更新公式中涉及的五个元素:状态(State)、动作(Action)、奖励(Reward)、下一状态(NextState)、下一动作(NextAction),即(S,A,R,S’,A’)。SARSA与Q-Lear
- 因果推断推荐系统工具箱 - PRS(二)
processor4d
文章名称【WSDM-2021】【UniversityofVirginia-Google】Non-ClicksMeanIrrelevant?PropensityRatioScoringAsaCorrection核心要点上一节讲解了在unbiasL2R的场景中,基于pairwise比较的损失函数的IPS的方法存在与真实评估指标偏离的问题,这一节讲解如何环节这一问题,并学习模型参数。方法细节问题引入作者
- centos9 配置yum源(清华源)
风异
centos
sudocp-r/etc/yum.repos.d/etc/yum.repos.d.backup备份yum源sudovim/etc/yum.repos.d/CentOS-Base.repo编辑yum源仓库[baseos]name=CentOSStream$releasever-BaseOSbaseurl=https://mirrors.tuna.tsinghua.edu.cn/centos-stre
- 【YOLO系列】YOLOv1详解:模型结构、损失函数、训练方法及代码实现
一碗白开水一
yolo系列助你拿捏AI算法YOLO人工智能目标检测计算机视觉
YOLOv1(YouOnlyLookOnce):实时目标检测的革命性突破✨motivation在目标检测领域,传统方法如R-CNN系列存在计算冗余、推理速度慢的问题。2016年提出的YOLO(YouOnlyLookOnce)首次实现端到端单阶段检测,将检测速度提升至45FPS(FasterR-CNN仅7FPS),彻底改变了实时目标检测的格局。其核心思想是将检测视为回归问题,实现"看一眼即知全貌"的
- 初始化 K8s 主节点时 报错failed to pull image registry.aliyuncs.com/google_containers/kube-apiserver:v1.23.17
Ashmcracker
kubernetes容器云原生
运行r如下命令初始化kubernetes的master节点2025年3月12日更新:阿里云的镜像仓库目前只给它自己云上的服务器使用了,建议更换华为云的镜像加速器https://support.huaweicloud.com/usermanual-swr/swr_01_0045.htmlkubeadminit\--kubernetes-version=v1.23.17\--image-reposit
- R for data science翻译笔记1.1 introduction
七月0317
MNE-Python翻译版-中文官方文档笔记信息可视化
本书第一章节(1.1-1.8)的目标是让读者尽快掌握数据探索的基本工具。数据探索是查看数据、快速生成假设、快速测试,然后不断重复的艺术。数据探索的目标是生成许多有希望的线索,我们可以稍后进行更深入的探索。在本书的这一部分中,你将学习一些有用的工具,它们可以立即带来回报:可视化是R编程的绝佳起点,因为反馈非常明显:你可以绘制优雅且信息量大的图形来帮助你理解数据。在数据可视化中,您将深入可视化,学习g
- R拟合 | 一个分布能看到三个峰,怎么拟合出这三个正态分布的参数? | 高斯混合模型 与 EM算法
biomooc
R数学与统计r语言
1.效果已知数据符合上图分布,怎么求下图的三个分布的参数mu,sigma,及每个分布的权重lambda?2.代码:高斯混合模型(GaussianMixtureModel,简称GMM)library(mixtools)set.seed(123)#确保结果可重复#假设x是你的观测数据xsummary(mix)summaryofnormalmixEMobject:comp1comp2comp3lambd
- 《R for Data Science (2e)》免费中文翻译 (第2章) --- Workflow: basics
TigerZ 生信宝库
r语言数据科学中文翻译
写在前面本系列推文为《RforDataScience(2)》的中文翻译版本。所有内容都通过开源免费的方式上传至Github,欢迎大家参与贡献,详细信息见:Books-zh-cn项目介绍:Books-zh-cn:开源免费的中文书籍社区r4ds-zh-cnGithub地址:https://github.com/Books-zh-cn/r4ds-zh-cnr4ds-zh-cn网站地址:https://b
- 读书笔记《道德经说什么》38
和弦夜光杯
原文:上德不德,是以有德;下德不失德,是以无德。上德无为而无以为,下德无为而有以为;上仁为之而无以为;上义为之而有以为;上礼为之而莫之应,则攘臂而扔之。故失道而后德,失德而后仁,失仁而后义,失义而后礼。夫礼者,忠信之薄而乱之首;前识者,道之华而愚之始。是以大丈夫处其厚,不居其薄;处其实,不居其华。故去彼取此。R十行采集:1.上德不德,是以有德:最有德的人,做了有德的事儿,没有觉得有什么了不起。上德
- 《刻意学习》2/7-2组-阿静-闲杂人
闲杂人_3d45
R原文拆页你可以看看自己每一天,有多少时间,本来应该遵守规则,却在拿灵活性当挡箭牌。所谓“规则”,既可以是社会制定的,也可以是自己制定的。比如自己说每天晚上都要去跑步,这是你给自己定的规则,然后某一天突然懒劲犯了,于是告诉自己,今天灵活调整一天。于是就没有遵守规则,用灵活性来“补位”。当然反对者认为,每天跑步很辛苦,人是要休息的,所以怎么能没有灵活性呢?这个观点从理论上讲是没错,但是在思考逻辑上却
- java的break能加标签,return可以加标签吗
悟能不能悟
java开发语言
在Java中,return语句不能像break那样使用标签。这是由两者的设计目的决定的:关键区别:**break+标签用于跳出指定的循环/代码块**(如多层嵌套循环),在需要从深层嵌套中直接退出外层时非常有用:outerLoop:for(inti=0;i<10;i++){for(intj=0;j<10;j++){if(condition)breakouterLoop;//直接跳出外层循环}}**r
- LeetCode[位运算] - #137 Single Number II
Cwind
javaAlgorithmLeetCode题解位运算
原题链接:#137 Single Number II
要求:
给定一个整型数组,其中除了一个元素之外,每个元素都出现三次。找出这个元素
注意:算法的时间复杂度应为O(n),最好不使用额外的内存空间
难度:中等
分析:
与#136类似,都是考察位运算。不过出现两次的可以使用异或运算的特性 n XOR n = 0, n XOR 0 = n,即某一
- 《JavaScript语言精粹》笔记
aijuans
JavaScript
0、JavaScript的简单数据类型包括数字、字符创、布尔值(true/false)、null和undefined值,其它值都是对象。
1、JavaScript只有一个数字类型,它在内部被表示为64位的浮点数。没有分离出整数,所以1和1.0的值相同。
2、NaN是一个数值,表示一个不能产生正常结果的运算结果。NaN不等于任何值,包括它本身。可以用函数isNaN(number)检测NaN,但是
- 你应该更新的Java知识之常用程序库
Kai_Ge
java
在很多人眼中,Java 已经是一门垂垂老矣的语言,但并不妨碍 Java 世界依然在前进。如果你曾离开 Java,云游于其它世界,或是每日只在遗留代码中挣扎,或许是时候抬起头,看看老 Java 中的新东西。
Guava
Guava[gwɑ:və],一句话,只要你做Java项目,就应该用Guava(Github)。
guava 是 Google 出品的一套 Java 核心库,在我看来,它甚至应该
- HttpClient
120153216
httpclient
/**
* 可以传对象的请求转发,对象已流形式放入HTTP中
*/
public static Object doPost(Map<String,Object> parmMap,String url)
{
Object object = null;
HttpClient hc = new HttpClient();
String fullURL
- Django model字段类型清单
2002wmj
django
Django 通过 models 实现数据库的创建、修改、删除等操作,本文为模型中一般常用的类型的清单,便于查询和使用: AutoField:一个自动递增的整型字段,添加记录时它会自动增长。你通常不需要直接使用这个字段;如果你不指定主键的话,系统会自动添加一个主键字段到你的model。(参阅自动主键字段) BooleanField:布尔字段,管理工具里会自动将其描述为checkbox。 Cha
- 在SQLSERVER中查找消耗CPU最多的SQL
357029540
SQL Server
返回消耗CPU数目最多的10条语句
SELECT TOP 10
total_worker_time/execution_count AS avg_cpu_cost, plan_handle,
execution_count,
(SELECT SUBSTRING(text, statement_start_of
- Myeclipse项目无法部署,Undefined exploded archive location
7454103
eclipseMyEclipse
做个备忘!
错误信息为:
Undefined exploded archive location
原因:
在工程转移过程中,导致工程的配置文件出错;
解决方法:
- GMT时间格式转换
adminjun
GMT时间转换
普通的时间转换问题我这里就不再罗嗦了,我想大家应该都会那种低级的转换问题吧,现在我向大家总结一下如何转换GMT时间格式,这种格式的转换方法网上还不是很多,所以有必要总结一下,也算给有需要的朋友一个小小的帮助啦。
1、可以使用
SimpleDateFormat SimpleDateFormat
EEE-三位星期
d-天
MMM-月
yyyy-四位年
- Oracle数据库新装连接串问题
aijuans
oracle数据库
割接新装了数据库,客户端登陆无问题,apache/cgi-bin程序有问题,sqlnet.log日志如下:
Fatal NI connect error 12170.
VERSION INFORMATION: TNS for Linux: Version 10.2.0.4.0 - Product
- 回顾java数组复制
ayaoxinchao
java数组
在写这篇文章之前,也看了一些别人写的,基本上都是大同小异。文章是对java数组复制基础知识的回顾,算是作为学习笔记,供以后自己翻阅。首先,简单想一下这个问题:为什么要复制数组?我的个人理解:在我们在利用一个数组时,在每一次使用,我们都希望它的值是初始值。这时我们就要对数组进行复制,以达到原始数组值的安全性。java数组复制大致分为3种方式:①for循环方式 ②clone方式 ③arrayCopy方
- java web会话监听并使用spring注入
bewithme
Java Web
在java web应用中,当你想在建立会话或移除会话时,让系统做某些事情,比如说,统计在线用户,每当有用户登录时,或退出时,那么可以用下面这个监听器来监听。
import java.util.ArrayList;
import java.ut
- NoSQL数据库之Redis数据库管理(Redis的常用命令及高级应用)
bijian1013
redis数据库NoSQL
一 .Redis常用命令
Redis提供了丰富的命令对数据库和各种数据库类型进行操作,这些命令可以在Linux终端使用。
a.键值相关命令
b.服务器相关命令
1.键值相关命令
&
- java枚举序列化问题
bingyingao
java枚举序列化
对象在网络中传输离不开序列化和反序列化。而如果序列化的对象中有枚举值就要特别注意一些发布兼容问题:
1.加一个枚举值
新机器代码读分布式缓存中老对象,没有问题,不会抛异常。
老机器代码读分布式缓存中新对像,反序列化会中断,所以在所有机器发布完成之前要避免出现新对象,或者提前让老机器拥有新增枚举的jar。
2.删一个枚举值
新机器代码读分布式缓存中老对象,反序列
- 【Spark七十八】Spark Kyro序列化
bit1129
spark
当使用SparkContext的saveAsObjectFile方法将对象序列化到文件,以及通过objectFile方法将对象从文件反序列出来的时候,Spark默认使用Java的序列化以及反序列化机制,通常情况下,这种序列化机制是很低效的,Spark支持使用Kyro作为对象的序列化和反序列化机制,序列化的速度比java更快,但是使用Kyro时要注意,Kyro目前还是有些bug。
Spark
- Hybridizing OO and Functional Design
bookjovi
erlanghaskell
推荐博文:
Tell Above, and Ask Below - Hybridizing OO and Functional Design
文章中把OO和FP讲的深入透彻,里面把smalltalk和haskell作为典型的两种编程范式代表语言,此点本人极为同意,smalltalk可以说是最能体现OO设计的面向对象语言,smalltalk的作者Alan kay也是OO的最早先驱,
- Java-Collections Framework学习与总结-HashMap
BrokenDreams
Collections
开发中常常会用到这样一种数据结构,根据一个关键字,找到所需的信息。这个过程有点像查字典,拿到一个key,去字典表中查找对应的value。Java1.0版本提供了这样的类java.util.Dictionary(抽象类),基本上支持字典表的操作。后来引入了Map接口,更好的描述的这种数据结构。
&nb
- 读《研磨设计模式》-代码笔记-职责链模式-Chain Of Responsibility
bylijinnan
java设计模式
声明: 本文只为方便我个人查阅和理解,详细的分析以及源代码请移步 原作者的博客http://chjavach.iteye.com/
/**
* 业务逻辑:项目经理只能处理500以下的费用申请,部门经理是1000,总经理不设限。简单起见,只同意“Tom”的申请
* bylijinnan
*/
abstract class Handler {
/*
- Android中启动外部程序
cherishLC
android
1、启动外部程序
引用自:
http://blog.csdn.net/linxcool/article/details/7692374
//方法一
Intent intent=new Intent();
//包名 包名+类名(全路径)
intent.setClassName("com.linxcool", "com.linxcool.PlaneActi
- summary_keep_rate
coollyj
SUM
BEGIN
/*DECLARE minDate varchar(20) ;
DECLARE maxDate varchar(20) ;*/
DECLARE stkDate varchar(20) ;
DECLARE done int default -1;
/* 游标中 注册服务器地址 */
DE
- hadoop hdfs 添加数据目录出错
daizj
hadoophdfs扩容
由于原来配置的hadoop data目录快要用满了,故准备修改配置文件增加数据目录,以便扩容,但由于疏忽,把core-site.xml, hdfs-site.xml配置文件dfs.datanode.data.dir 配置项增加了配置目录,但未创建实际目录,重启datanode服务时,报如下错误:
2014-11-18 08:51:39,128 WARN org.apache.hadoop.h
- grep 目录级联查找
dongwei_6688
grep
在Mac或者Linux下使用grep进行文件内容查找时,如果给定的目标搜索路径是当前目录,那么它默认只搜索当前目录下的文件,而不会搜索其下面子目录中的文件内容,如果想级联搜索下级目录,需要使用一个“-r”参数:
grep -n -r "GET" .
上面的命令将会找出当前目录“.”及当前目录中所有下级目录
- yii 修改模块使用的布局文件
dcj3sjt126com
yiilayouts
方法一:yii模块默认使用系统当前的主题布局文件,如果在主配置文件中配置了主题比如: 'theme'=>'mythm', 那么yii的模块就使用 protected/themes/mythm/views/layouts 下的布局文件; 如果未配置主题,那么 yii的模块就使用 protected/views/layouts 下的布局文件, 总之默认不是使用自身目录 pr
- 设计模式之单例模式
come_for_dream
设计模式单例模式懒汉式饿汉式双重检验锁失败无序写入
今天该来的面试还没来,这个店估计不会来电话了,安静下来写写博客也不错,没事翻了翻小易哥的博客甚至与大牛们之间的差距,基础知识不扎实建起来的楼再高也只能是危楼罢了,陈下心回归基础把以前学过的东西总结一下。
*********************************
- 8、数组
豆豆咖啡
二维数组数组一维数组
一、概念
数组是同一种类型数据的集合。其实数组就是一个容器。
二、好处
可以自动给数组中的元素从0开始编号,方便操作这些元素
三、格式
//一维数组
1,元素类型[] 变量名 = new 元素类型[元素的个数]
int[] arr =
- Decode Ways
hcx2013
decode
A message containing letters from A-Z is being encoded to numbers using the following mapping:
'A' -> 1
'B' -> 2
...
'Z' -> 26
Given an encoded message containing digits, det
- Spring4.1新特性——异步调度和事件机制的异常处理
jinnianshilongnian
spring 4.1
目录
Spring4.1新特性——综述
Spring4.1新特性——Spring核心部分及其他
Spring4.1新特性——Spring缓存框架增强
Spring4.1新特性——异步调用和事件机制的异常处理
Spring4.1新特性——数据库集成测试脚本初始化
Spring4.1新特性——Spring MVC增强
Spring4.1新特性——页面自动化测试框架Spring MVC T
- squid3(高命中率)缓存服务器配置
liyonghui160com
系统:centos 5.x
需要的软件:squid-3.0.STABLE25.tar.gz
1.下载squid
wget http://www.squid-cache.org/Versions/v3/3.0/squid-3.0.STABLE25.tar.gz
tar zxf squid-3.0.STABLE25.tar.gz &&
- 避免Java应用中NullPointerException的技巧和最佳实践
pda158
java
1) 从已知的String对象中调用equals()和equalsIgnoreCase()方法,而非未知对象。 总是从已知的非空String对象中调用equals()方法。因为equals()方法是对称的,调用a.equals(b)和调用b.equals(a)是完全相同的,这也是为什么程序员对于对象a和b这么不上心。如果调用者是空指针,这种调用可能导致一个空指针异常
Object unk
- 如何在Swift语言中创建http请求
shoothao
httpswift
概述:本文通过实例从同步和异步两种方式上回答了”如何在Swift语言中创建http请求“的问题。
如果你对Objective-C比较了解的话,对于如何创建http请求你一定驾轻就熟了,而新语言Swift与其相比只有语法上的区别。但是,对才接触到这个崭新平台的初学者来说,他们仍然想知道“如何在Swift语言中创建http请求?”。
在这里,我将作出一些建议来回答上述问题。常见的
- Spring事务的传播方式
uule
spring事务
传播方式:
新建事务
required
required_new - 挂起当前
非事务方式运行
supports
&nbs