基于深度学习的腹部CT的肝脏肿瘤分割的项目笔记
一、前言:
- 1
这个肝脏肿瘤分割项目是我们大二下学期(2020年1月开始)申请的一个大创项目。
此前我有参与过一个关于深度学习的项目(基于深度学习的视频识别),但在这个项目中我算是摸摸鱼的角色,but 也还是有收获的啦!比如:对做这些项目的流程算是有一个大致的了解 ? 打开了深度学习的门。。。
- 2
写这篇文章就是想记录一下我做第二个项目的历程。(因为今天10.24,又在晚上刚好把代码调通了!!!真的是过节了吖!!!所以就写个回忆散记来庆祝这激动人心的时刻嘻嘻~)
二、关于肝项目的经历和想法:
总体来说,项目的过程其实是不太顺畅的。。。现在凭借我感人的记忆力按时间顺序回忆一些难忘的过程节点。(不想看太多我这平凡琐碎的经历的话,可以直接看最后精华的两点噢~哈哈哈)
- 1
首先,一开始我们还没想到用云服务器来跑代码(算是走了点弯路)。所以,之后在尝试跑别人的代码的时候,由于本地环境配置的问题,异常艰难!!!好吧,就是一次都没跑通过。环境问题一直有报错,这就费了我们几个月时间,现在想起来,真的太费时间!
- 2
在项目整个前半段(有六七个月的时间了),我们是在网上找各种资料(收藏了等于看了 )在知网阅读文献、在CSDN看相关项目的文章、在知乎看大佬的医学图像处理的专栏文章、还有在github上查找和学习相关代码等等。
- 3
在前期找资料时,在CSDN发现了一篇博客,偶买噶!!这不就是我们想要的项目指导书吗?
就是这篇:医学图像分割 基于深度学习的肝脏肿瘤分割 实战(一)_normol的博客-CSDN博客
这文章写得太详细啦!小白真是太感动了!第一篇是分割肝脏的,然后还有第二篇是分割肿瘤的。
我们想着就按照这两篇博客来完成这个项目。但是美好的想法还是和现实进展有点距离的。在这篇博客下面的评论中收获到很多有用的信息,并且下载了源代码,但是,由于前面说的,当时是用本地的pycharm来跑代码的,就一言难尽(由于环境的配置问题,一直有各种奇奇怪怪的报错),心态逐渐崩了昂。但是也是有认真学习了博主的方法和代码。印象中应该是花了一两个月来研究和尝试跑通这份代码的,但是很可惜并没有成功。。。
期间,在评论区的一位大佬帮助过程中,我了解到了百度的深度学习框架飞桨PaddlePaddle-源于产业实践的开源深度学习平台
- 4
接着,九月一号一开学就要进行项目中期汇报了。但是我们的项目仿佛没有实质性的成果。噢!我的老天鹅,这就有点尴尬了。
本着毛主席说的“东方不亮西方亮”,我们尝试找其他参考资料。然后又发现了一篇博客!!Tensorflow 2.0 医学图像分割(肝脏CT图像分割)_qq_38412266的博客-CSDN博客
(并且这个博主小哥太nice啦,给我很多帮助!真是十分感谢!!)
但是呢,这份代码只是将腹部CT图的肝脏分割出来了,肿瘤部分还得再搜寻搜寻。。。然后在博主大佬的帮助下,我学了用kaggle云服务器来跑代码。在kaggle上面还有很多有趣的开源项目,之后有时间可以运行玩一下噢~
- 5
然后就在十月初的时候,我找到了两份github上的代码。(但是现在我忘了当时是在什么机缘偶合的情况下找到的。。。找的时候,我也会顺着他们github的个人主页来看一下有没有相关的代码~)
https://github.com/zz10001/Thesis-LiTS2017/blob/master/preprocessing.py
重点是下面这一份!!
Confusezius/unet-lits-2d-pipeline: Liver Lesion Segmentation with 2D Unets
这份代码,作者的README里面的步骤写的很详细!!我跑的就是这一份代码~ 这个README得认真看,看懂每一步(一开始我以为他的大小Net都要运行,之后才觉悟二选一就行)
一开始一直都是用kaggle的云服务器,但是后来中途报错显示爆内存了(kaggle和colab都有13G内存好像)。然后就转到去谷歌的Colab了。
这也是我第一次用Google Colab,百度一下怎么用,还是很快上手的噢~不得不说真的太赞了!体验很好吖,用着很顺手哈哈哈。(我是之前在干第一个视频识别项目的时候,注册了谷歌的账号)Google Colab——用谷歌免费GPU跑你的深度学习代码
之后,在colab我就跟着README里面的步骤,敲命令来运行的,某个代码有报错的话,就改了代码再重新把这个.py上传到谷歌的云端硬盘(不能直接在云盘改的噢),虽然感觉反复上传更新很复杂,其实也没有很复杂噢,上传py文件还是很快的。感觉代码主要是改一下路径,报错也不是很牛角尖的错,在stack overflow都有方法~
然鹅,就在过节的今天(1024),代码全部调通了哈哈哈!!
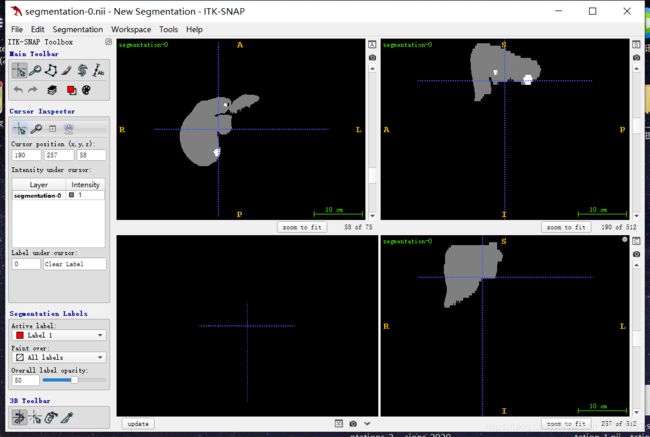
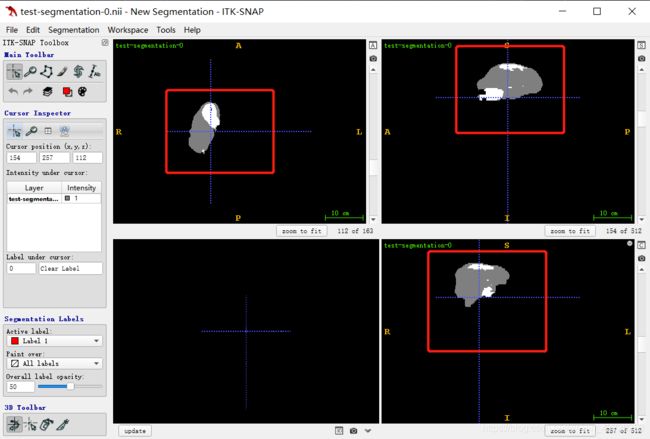
但是有个问题就是,我就上传了三四个nii文件作为数据集(因为怕内存不够),所以训练出来的效果不太行。肝脏分割的DICE参数是0.91(还不错噢),但是肿瘤分割的DICE参数只有0.16,所以出来的结果是一大片肿瘤区域。
下面两幅图,第一个是原数据集中的标准肝脏和白色的肿瘤,第二个是训练出来的分割结果。(我是用ITK-SNAP来打开nii文件的)
现在项目算是完成百分之六七十啦~
下一步试一下加多nii数据的数量来训练~
二、结语
emmm 这项目笔记就写到这儿叭,很遗憾,这虎头蛇尾的。
很抱歉不能一一回复私信,因为我可能有心无力去解答。。。
嗐,之后也不读研,所以深度学习的学习可能就到此为止啦~~
(遗憾离场)