pymol安装使用;vscode蛋白质可视化插件 protein viewer;rcsb pdb,fasta蛋白wget下载
1、pymol安装使用
参考:https://blog.csdn.net/eternalapple/article/details/110263296
官网:https://pymol.org/
安装:
1、可以直接conda安装,一条命令
conda install -c schrodinger pymol-bundle
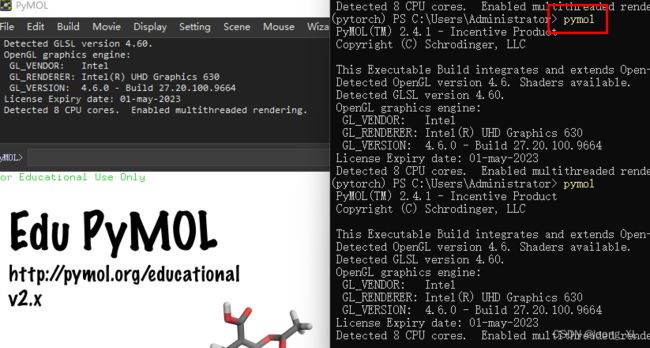
安装成功后输入pymol即可调出窗口
****激活码申请
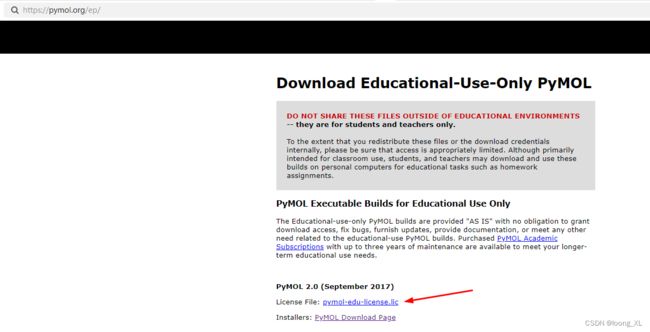
可以申请教育学生,可以不是学校邮箱一般也可以
申请成功后收到邮件,里面有链接账号密码,然后就可以下载许可
2、蛋白质可视化插件 protein viewer
这是vscode编辑器的插件,类似pymol软件展示
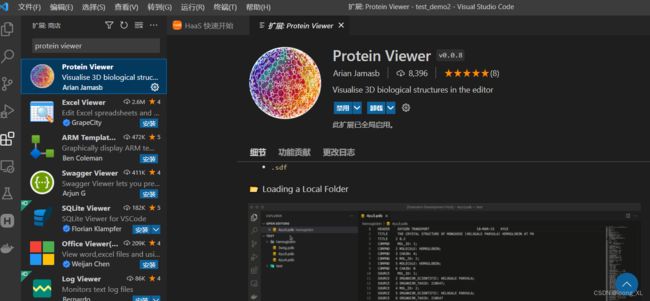
1) a Protein Structure from the PDB or AlphaFoldDB 支持pdb库蛋白搜索和本地文件导入
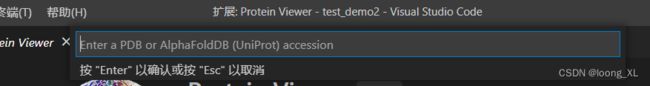
a、 ctrl +shift+ p 调出框输入 Start Protein Viewer ,命令后即可输入PDB or AlphaFoldDB 库的蛋白质id
.pdb
.pdbqt
.mmcif
.gro
.xyz
.cif
.mol
.mol2
.sdf
3、rcsb pdb蛋白wget下载
RCSB PDB (http://www.rcsb.org/) 蛋白质数据库PDB最早由美国布鲁克海文国家实验室建立
url = 'http://www.rcsb.org/pdb/files/%s.pdb.gz' % (pdb_id.upper()) ##下载蛋白路径
dest = '%s/%s.pdb.gz' % (os.path.abspath(dest_dir), pdb_id) #保存文件路径
wget --quiet %s -O %s' % (url, dest)
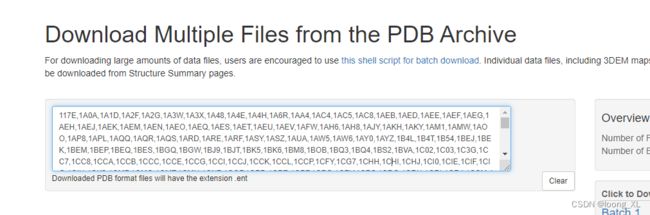
rcsb pdb批量下载
### 复制过来转化成列表
a11="117E,1A0A,1A1D,1A2F,1A2G,1A3W,1A3X,**********8EAV,8ESC,8PRK"
b11 = a11.split(",")
b11
## 循环下载
import requests
for pdb in b11:
r = requests.get(f"https://files.rcsb.org/download/{pdb}.pdb", stream=True)
f = open(f"F:\pdb_database\{pdb}.pdb", "wb")
for chunk in r.iter_content(chunk_size=512):
if chunk:
f.write(chunk)
f.close()
fasta 格式下载
for pdb in b11:
r = requests.get(f"https://www.rcsb.org/fasta/entry/{pdb}", stream=True) ##pdb这里更改蛋白id就行
f = open(f"F:\pdb_database\{pdb}.fasta", "wb")
for chunk in r.iter_content(chunk_size=512):
if chunk:
f.write(chunk)
f.close()