(二十五:2020.12.15)CVPR 2020 学习(三)《U^2 Net》训练实操(21.1.4更新)
《U^2 Net: Going Deeper with Nested U-Structure for Salient Object Detection》《借助嵌套U型结构深入研究显着物体》
- 讲在前面
- 一、如何整理你的医学图像数据集
-
- 1.选择数据集
- 2.转换你的data
- 3.转换你的label
- 4.将数据放置在对应的路径下
- 二、使用你整理好的数据进行训练
-
- 1.`ValueError: At least one stride in the given numpy array is negative, and tensors with negative strides are not currently supported. (You can probably work around this by making a copy of your array with array.copy().)`
- 2.`IndexError: invalid index of a 0-dim tensor. Use tensor.item() in Python or tensor.item() in C++ to convert a 0-dim tensor to a number`
- 3.3090的环境问题
- 三、执行`u2net_train.py`,跑起来!!!
- 四、测试训练完的模型的结果
-
- 1.先看下我的过拟合版本的结果
- 2.再看下我的其他版本的结果
- 3.看下nnUNet的模型推理结果
- 4.实验结论
-
- 1.nnUNet
- 2.u2net
讲在前面
- 一.因为推理和测试部分很简单,也有对应的模型下载,这里不做多的赘述;
- 二.由于作者用的torch是0.4.0,所以在实际操作的过程中,遇到一些版本兼容的问题,在这里我做一下记录,希望看了该博客大家能够顺利的使用U2-net。
- 三.我用了自己的肾脏数据集,因为原始数据是nii,所以我将数据转换成了2d的图片进行训练,这里自己的代码公开一下,方便各位医学图像处理的读者进行更方便的处理。
- 四.我的6000张图片已经开始3090的训练,让我们等待一个过拟合版本,看看是否真的配的上它如此高的热度。
- 五.之前的训练因为把训练集搞错了自己有点晕也一直没有发现,以为这个模型用不起,最近空下来发现这个问题,今天更新下我的训练结果。
一、如何整理你的医学图像数据集
1.选择数据集
我这里选择的是肾脏的数据集,网上公开的此类数据集有很多,但是很多都是nii文件,所以你需要将这些文件转成2d的图片格式。
我的数据集就是之前nnUNet的训练数据集
2.转换你的data
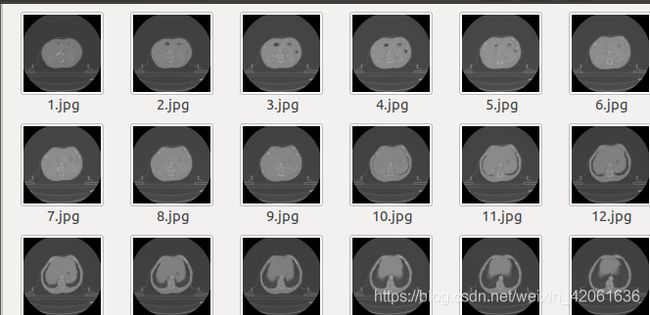
也就是之前nnUNet里面的imagesTr文件夹(未加_0000之前的那个文件夹),也就是这种:
我在这里用下面代码进行转换,这里转换为jpg文件:
nii_files = r'/media/qiao/WindowsData/u2net_test/nii_data/imagesTr_'
image_name = r'/media/qiao/WindowsData/u2net_test/nii_data/data'
filelist = os.listdir(nii_files)
filelist.sort(key=lambda x: int(x[-10:-7].zfill(3)))
h = 1
for f in filelist:
file = os.path.join(nii_files, f)
volume = sitk.ReadImage(file)
array = sitk.GetArrayFromImage(volume)
for i in range(array.shape[0]):
nii_slice = array[i, :, :]
imageio.imwrite(os.path.join(image_name, '{}.jpg'.format(h)), nii_slice)
h += 1
3.转换你的label
同样的数据看起来如下:
我在这里用下面代码进行转换,这里转换为png文件:
nii_files = r'/media/qiao/WindowsData/u2net_test/nii_data/labelsTr'
image_name = r'/media/qiao/WindowsData/u2net_test/nii_data/label'
filelist = os.listdir(nii_files)
filelist.sort(key=lambda x: int(x[-10:-7].zfill(3)))
h = 1
for f in filelist:
file = os.path.join(nii_files, f)
volume = sitk.ReadImage(file)
array = sitk.GetArrayFromImage(volume)
for i in range(array.shape[0]):
nii_slice = array[i, :, :]
imageio.imwrite(os.path.join(image_name, '{}.png'.format(h)), nii_slice)
h += 1
4.将数据放置在对应的路径下
像我这样创建文件夹,这里的gt_aug是之前的label文件夹,这里的im_aug是之前的data文件夹,这里面的gt_aug_255是我创建的文件夹,不用管:
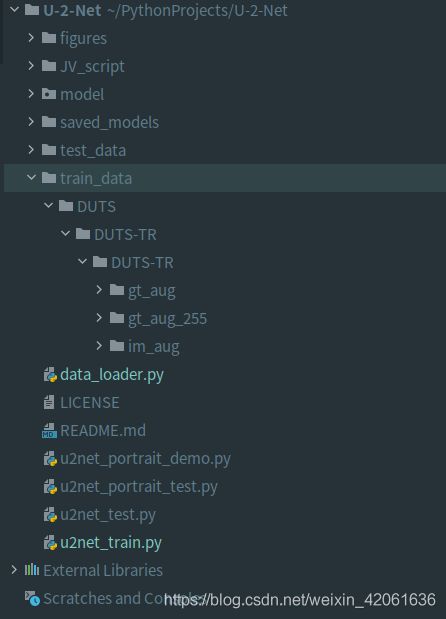
二、使用你整理好的数据进行训练
在上述步骤完成后,其实你只要执行u2net_train.py就行了,但是因为作者的版本太低,所以会遇到一些问题,大致解决方案如下:
1.ValueError: At least one stride in the given numpy array is negative, and tensors with negative strides are not currently supported. (You can probably work around this by making a copy of your array with array.copy().)
在data_loader.py中的第224行左右,改成下面这样,即后面加上.copy():
![]()
2.IndexError: invalid index of a 0-dim tensor. Use tensor.item() in Python or tensor.item() in C++ to convert a 0-dim tensor to a number
将所有设计到损失函数,类似于loss.data[0]这样的部分,后面的[0]去掉,大概在u2net_train.py的第42行改成如下:
3.3090的环境问题
在我的1070+cuda10.1+torch1.6.0上,可以轻松运行,但是强调一点:如果在3090上进行训练,必须装的是1.8.0的torch和cuda11以上。官方的无脑pip安装并不行,需要从源编译或者直接装whl文件,这里有一个教程,我还没有试过。
三、执行u2net_train.py,跑起来!!!
寡人这就看代码去了!
四、测试训练完的模型的结果
1.先看下我的过拟合版本的结果
我选择的模型是下图中的最后一个,明显训练的损失开始波动,说明改模型已经过拟合。
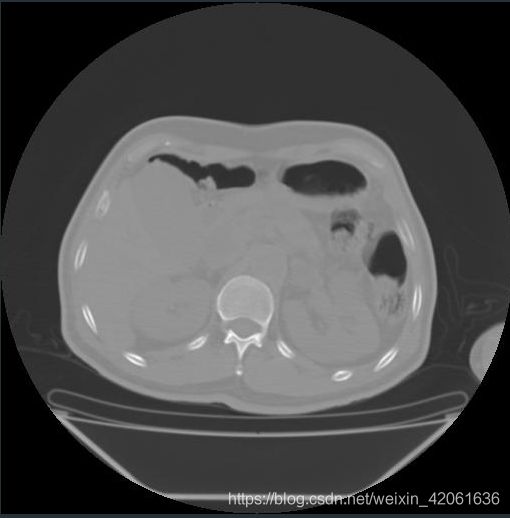
该模型为双肾模型,我测试了一个病例的数据,是全新的来自另一个医院的数据,在人眼观测到的比较明显的双肾的图像上,原图与结果测试如下:

 但是由于该模型过拟合且为2d的模型,所以在一些没有肾的图像上依然会有一些产生的噪声,这是后面持续优化的过程,我再来测试一下一个之前的模型。
但是由于该模型过拟合且为2d的模型,所以在一些没有肾的图像上依然会有一些产生的噪声,这是后面持续优化的过程,我再来测试一下一个之前的模型。
2.再看下我的其他版本的结果
选一个损失降到0.01的模型,结果显示:


边界稍微有点模糊,原因是模型的推理结果其实是一个三通道的图片,需要二值化或者修改网络拓扑的输出让他生成单通道的二值图。
3.看下nnUNet的模型推理结果
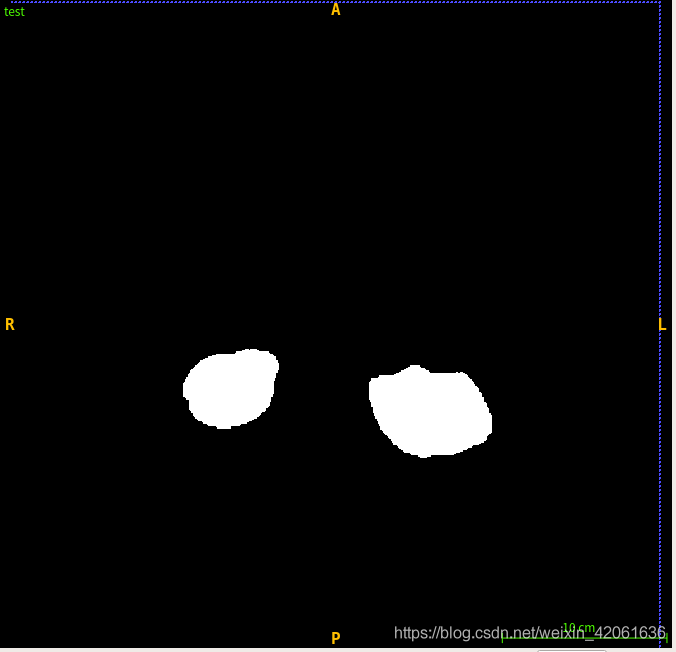
 可以看出nnUNet的结果依然不错。
可以看出nnUNet的结果依然不错。
4.实验结论
1.nnUNet
nnUNet的优点可以说是很明显,因为调用了3dunet的框架,所以不会存在2dunet的一些问题,对于上下文信息的处理不会在不该出现器官的层面上出现一些噪声和阴影。同时,表现出与标签的绝对一致性,只要你的标签足够准确和优秀,那么模型的精度也会相当可靠。
但nnUNet的缺点也很明显,推理时间慢的无法忍受,频繁的进行插值更是延长了推理的时间,我最近的工作将插值时间进行了急剧的缩减,有兴趣的同学可以留言给我。我也在github上告知了作者这方面的改进,但是搞学术的似乎对于速度的急速提升并不感冒。虽然如此,我仍然将真正实际的部署视为更重要的事情,我坚信,技术无法实现真正对人类的帮助那将毫无疑问。
2.u2net
对于这个以制作人物肖像画为目的的网络,我终于看到了其在医学影响影像的一点可利用价值。很明显,由于本身是2d网络,所以推理速度相当快。结果的显示,一个经过合适训练的模型,似乎也能具备一定的潜力。尤其这种平滑边缘的能力,是我相当欣赏的一点。
那么,加入u2net与nnunet结合去分割一些诸如胰腺这样边界模糊的困难任务,是否能改善其精度呢?我认为可以,因为从u2net的结构上来说,它所提取到的信息更多,编码器和解码器的信息置换是一种行之有效的方法。对u2net进行3d改进同时进行nnUNet移植将会是未来的工作。